马瑞丰,张威
分子生物学是目前自然学科中进展最迅速、最具活力和生气的领域之一。借助于分子生物学的发展,人类已经解决了许多涉及生物的进化、运行机理等重大科学问题。将分子生物学技术与传统古气候学、古生态学等学科交叉和融合,有望在现有的地球科学研究中碰撞出新的火花。
一、沉积 植物aDNA的定义及年代
沉积植物aDNA属于古环境DNA的范畴,aDNA主要是机体死亡经历水解和氧化后、保存在沉积物中的DNA分子基因物质。在植物中,这些aDNA分子主要赋存于植物残体组织,如枝干、果实和种子的厚壁组织及植物化石残体中(图1)。这些埋藏于沉积物中的古植物来源的DNA就是沉积植物aDNA(Felix et al., 2010)。沉积植物aDNA是我们分析古植被信息的主要手段,也是近十年来应用非常广泛的古植被重建方法。

图1 沉积物中分离出的植物残体
大量研究显示,沉积植物aDNA最老记录已超过五万五千年,而冰芯可将其保存长达45-80万年(Eske et al., 2014),孢粉中的aDNA为15万年(Suyama, et al., 1996)。基于以上植物aDNA的保存时间,可完全实现通过沉积植物aDNA的信息来重建晚更新世及全新世的植被演替过程。

图2 沉积过程与古植被重建图示
二、沉积植物aDNA的来源
研究表明,湖泊沉积物中的孢粉来自于区域的植物源区,且以风媒植物孢粉为主(Wilmshurst et al., 2005; Chen et al., 2017)。此类孢粉通过气流可实现区域性分布,其传播距离要远高于虫媒植物孢粉。由于孢粉可进行风力传输,记录了较大区域性的植物区系。由于植物残体体积重量较大,不易被风力搬运(Allen and Huntley., 2010),因此植物大化石来自于本地植物,其种类通常受湖泊周围水生和湿地植物种类影响,陆生植物代表性不足(Birks., 2007)。相关研究证实,沉积物中的植物aDNA与沉积孢粉单体颗粒中的DNA并不完全一致,其与植物大化石有着相似的来源(Pedersen., 2013; Lendvay et al., 2018),其具体关系见图1。大量研究表明(Barnes and Turner., 2016; Ficetola et al.,2015; Yoccoz., 2012; Boessenkool et al., 2014),aDNA技术虽不能完全体现植被调查以及孢粉鉴定的数据,但其有较大范围的重合,对于植物大化石的种类可基本覆盖。
Metabarcoding和Shotgun metabarcoding是两种目前发展最为迅速的DNA宏条形码技术。这些技术主要通过环境DNA来评估生物多样性,其利用PCR和第二代测序等分子技术,结合经典生态学知识,通过生物信息学和生物统计学分析来实现生物多样性的监测。Shotgun metabarcoding有着更高检测精度,可对更多的种类进行覆盖。
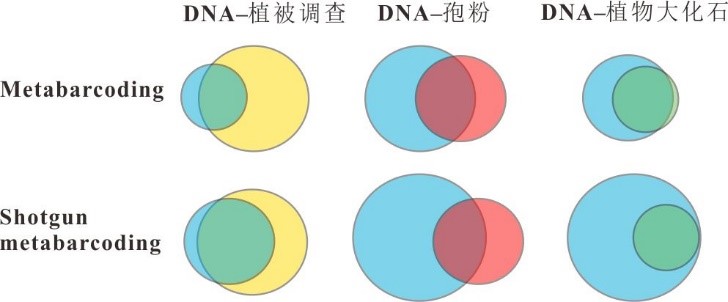
图3 基于3种方法重建的植物种类比例韦恩图(蓝色为两种aDNA技术,黄色为植被调查,红色为孢粉统计,绿色为植物大化石)
三、沉积植物aDNA采样分析要点
植物aDNA沉积物岩芯的收集与常规古生态研究中的钻取设备一致,但采样管需提前进行杀菌消毒处理,以杜绝现代环境残留物质对样品的污染。野外样品需要通过密封管来将样品运到实验室。样品处理及分析的各个阶段都应采取特别的预防措施。可能的污染来自于三个方面:设备本身、样品之间交叉污染以及实验药品。因此,每一个样品的分样都需要使用乙醇及短波紫外线(UVC)进行杀菌。消毒设备,保证无菌的工具是首要的,实验者在整个实验过程需确保穿着隔离实验服、口罩与手套要佩戴齐全。

图4 常规湖泊沉积物钻取过程
实验样品的选取需是样品芯最中心的部分。取样时候可以将DNA示踪剂用到取心设备上,这样就可以检测来自外层的DNA分子是否进入沉积物内部。防止外源DNA污染需要在整个实验过程严格把控。

图5 无菌操作台与岩心实验样品的选取
四、沉积植物aDNA的应用历史和进展
新技术的开启可以解决传统方法所不能解决的问题。沉积植物aDNA的研究始于植物孢粉DNA:德国学者Parducci(2010; 2012; 2014)率先从瑞典霍尔特贾宁冰湖(Holtjärnen postglacial lake)沉积物中提取到了苏格兰松树和挪威云杉的花粉质体DNA,这些花粉有近10万年的历史,说明植物孢粉DNA在10万年以内可以得到很好的保存。随后,在斯堪的纳维亚采用aDNA技术发现了保存于1.03万年的本地特有云杉属的DNA,2.2万年和17.7万年的松属及云杉属的叶绿体DNA;在斯堪的纳维亚中部通过两个全新世湖泊样品的采集,发现孢粉与aDNA鉴定科属上存在一定差异,并在孢粉统计到了46个科属,植物aDNA技术识别到了14个科属,二者综合统计了52个科属。从此,运用沉积植物aDNA技术对于古植被重建是一个重要的补充。
(1)提升对植物种类的识别
Epp et al(2015)在格陵兰北部湖泊沉积物中采用aDNA手段发现了冰后期维管植物,如岩高兰(Empetrum nigrum)等,并认为基于aDNA重建的植被与预期一致,可准确跟踪环境变化。德国学者Zimmermann et al(2017)在西伯利亚东北部Buor Khaya半岛沉积物中,通过分析沉积aDNA鉴定出了154个科属的植物,孢粉统计出了83个科属,二者共同重建了该区域末次冰期之后的气候,并认为在沉积剖面中,aDNA表现为高分辨率高物种检测丰度的特点,并指出沉积aDNA的种类更多的表现了植物地下生物量的信息。
(2)补充植物大化石不足造成的信息缺失
在沉积体中缺乏植物大化石的情况下,采用沉积aDNA和孢粉结合的技术,具有较大优势。Alsos et al(2018)通过分析斯瓦尔巴特群岛湖泊沉积物,将aDNA技术与植物大化石鉴定对比,aDNA方法额外鉴定出了更多的植物种类:6种维管束植物、2种藻类及12种苔藓类,且每份样品中检测到的种类都要多于大化石(图6,图7);未保存大化石的时期,植被情况同样可识别且变化不大。其研究表明,植物群在古气候变化中具有一定的“韧性”,气温变动在2℃之内的情况下植被不会有较大的变动。

图6 基于aDNA技术重建的孢粉谱

图7 基于植物大化石重建的孢粉谱
Zale et al.(2018)在斯堪的纳维亚威赫塞尔(Weichselian)后期冰盖附近的冰川瓯穴中采用了微体古生物化石、aDNA、孢粉及硅藻分析,恢复了早期冰上植被系统,认为灌木(柳属,桦木属,杜鹃花科)以及落叶松属等植物均存在于末次冰消期。
(3)揭示更清晰的植被生态关系
Heineck et al.(2017)通过沉积aDNA技术结合碳氮元素比及同位素(δ13C, δ15N)研究了帕米尔高原Karakul湖2.9 万年以来水下植物组成生物量变化,清晰展示了水位变动与古植物多样性的关系。
(4)助力于科学新发现
Bremond et al.(2017)提取了非洲西部Sele湖沉积DNA,进一步论证了热带地区古DNA存在的可能并发现了22科21属的植物,该结果与孢粉结果重叠性很小,分类对本地植物指示性较强(图8)。还鉴定到了外来物种地瓜。可见此技术除了可以较强针对性的重建本地植被还可用于早期农业耕作历史方面的研究。
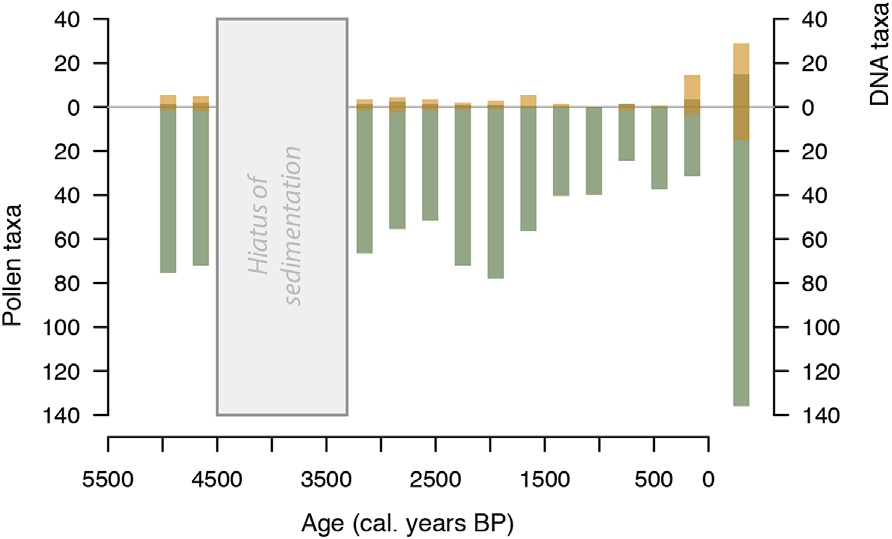
图8 DNA分类(黄色)与孢粉分类(绿色)对比图
Clarke et al.(2018)报道了Varanger半岛中部(挪威东北部)基于孢粉及沉积aDNA重建的植被及气候,共统计了101个维管植物和17个苔藓植物科属,其86%的科属依然存在于现在植被中,表明现代灌木-冻原群落及其大部分物种早在1.07万年前就已经存在,而aDNA结果特别显示了水生植物水芒草(Limosella aquatica)和杜鹃(Rhododendron tomentosum)的持续存在,说明整个全新世早期和中期的气候都要比现在温暖,这是依靠传统孢粉所记录不到的气候信息。
由此可见,高分辨率地挖掘沉积物中保存的植物化石与分子记录对于古植被演替及环境重建是非常关键的。随着技术本身的发展强化,其检测精度进一步提高,检测信息也愈加丰富,沉积aDNA手段不仅仅协助揭示植被的局部信息,将进一步达到全面客观展示本地古植被的演替过程。
本文第一作者系辽宁师范大学博士研究生,第二作者为辽宁师范大学地理科学学院教授。本文属作者认识,相关问题交流可通过邮箱874061015@qq.com与本人联系。欲知更多详情,请进一步阅读下列参考文献。
参考文献
[1] Pedersen M W, Overballe-Petersen S, Ermini L, et al. Ancient and modern environmental DNA.[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2014, 370(1660): 20130383.
[2] Jørgensen T, Haile J, Möller P, et al. A comparative study of ancient sedimentary DNA, pollen and macrofossils from permafrost sediments of northern Siberia reveals long-term vegetational stability[J]. Molecular Ecology, 2012, 21(8): 1989-2003.
[3] Parducci L, Matetovici I, Fontana S L, et al. Molecular- and pollen-based vegetation analysis in lake sediments from central Scandinavia[J]. Molecular Ecology, 2014, 23(4): 3511-3524.
[4] Eva B, Davey M L, H?Vard K, et al. Fungal palaeodiversity revealed using high-throughput metabarcoding of ancient DNA from arctic permafrost[J]. Environmental Microbiology, 2013, 15(4): 1176-1189.
[5] Jã¸Rgensen T, Kjaer K H, Haile J, et al. Islands in the ice: detecting past vegetation on Greenlandic nunataks using historical records and sedimentary ancient DNA meta-barcoding[J]. Molecular Ecology, 2012, 21(8): 1980-1988.
[6] Epp L S, Gussarova G, Boessenkool S, et al. Lake sediment multi-taxon DNA from North Greenland records early post-glacial appearance of vascular plants and accurately tracks environmental changes[J]. Quaternary Science Reviews, 2015, 117(2): 152-163.
[7] Rawlence N J, Lowe D J, J.R, et al. Using palaeoenvironmental DNA to reconstruct past environments: Progress and prospects[J]. Journal of Quaternary Science, 2015, 29(7): 610-626.
[8] 马瑞丰, 张威, 刘亮, 杨蝉玉. 沉积植物古DNA技术在古植被重建中的应用[J]. 沉积学报, 2020, 38(6): 1179-1191. doi: 10.14027/j.issn.1000-0550.2019.121.







